1. Η φιλοσοφία των βάσεων (τοπικές, on-line).
Οι βάσεις δεδομένων της SILVER
PLATTER είναι εγκατεστημένες τοπικά (στον database server) της Βιβλιοθήκης του
ΓΠΑ και προσβάσιμες μέσα από το τοπικό διαδίκτυο. Για την πρόσβαση σε αυτές
απαιτείται ένας κωδικός εισόδου που για την βιοτεχνολογία δίνεται παρακάτω και
ένα πρόγραμμα πρόσβασης διαδικτύου όπως Netscape ή Explorer. Υπάρχει και ειδικό
λογισμικό πρόσβασης (WΙΝSPIRS) για περισσότερο εξειδικευμένες χρήσεις. Η
πρόσβαση γίνεται στην διεύθυνση ( http://bibws4:8590/? ).
Η φιλοσοφία του λογισμικού πρόσβασης είναι ότι ο χρήστης μπορεί να αναζητήσει
πληροφορίες από μία ή και περισσότερες βάσεις ταυτόχρονα επιλέγοντας αρχικά
ποιές βάσεις από τις διαθέσιμες θέλει να συμπεριλάβει στις αναζητήσεις του. Με
ατό τον τρόπο και επιταχύνει τις αναζητήσεις του σε πολύπλευρα θέματα αλλά και
αυξάνει την πιθανότητα επιτυχίας της αναζήτησης σε β.δ. που προσεγγίζουν το
ίδιο θέμα από διαφορετικές θεματολογίες.
2. Ηλεκτρονική διεύθυνση ,
κωδικοί εισόδου.
Η αναζήτηση γίνεται στο site bibws4 της Βιβλιοθήκης του ΓΠΑ (http://bibws4:8590/?)
Κωδικοί χρηστών:
Ι. Για τo Webspirs
της Βιβλιοθήκης του ΓΠΑ (http://bibws4:8590/?)
Τμήμα Γεωπονικής
Βιοτεχνολογίας biotech
Κωδικοί εισόδου:
Ι. Για τo Webspirs
της Βιβλιοθήκης του ΓΠΑ (http://bibws4:8590/?)
Τμήμα Γεωπονικής Βιοτεχνολογίας biotech11
3. Το λογισμικό WEBSPIRS.
Το λογισμικό Webspirs προσφέρει
εύκολη και πολλαπλή πρόσβαση στις βάσεις δεδομένων της Silverplatter. Στο ΓΠΑ
είναι εγκατεστημένο στον εξυπηρετητή βάσεων δεδομένων της Βιβλιοθήκης και είναι
προσβάσιμο στην διεύθυνση ( http://bibws4:8590/? ) μέσω κάποιου προγράμματος πρόσβασης στο web (π.χ Netscape,
Explorer).
Με την είσοδο στο λογισμικό
απαιτείται η χρήση κωδικού και password που για την βιοτεχνολογία
δίνονται παραπάνω. Μετά την είσοδο στο λογισμικό γίνεται η επιλογή των
βάσεων δεδομένων που θα διερευνηθούν από τις διαθέσιμες στο ΓΠΑ. Μετά την
επιλογή το webspirs δίνει το παράθυρο απλής αναζήτησης. Για περισσότερο
πολύπλοκες αναζητήσεις υπάρχει η επιλογή build search. Οι όροι
αναζήτησης συνδέονται αρχικά με το και (and) αλλά μπορεί να γίνει και η
επιλογή του ή (or). Στην δεξιά πλευρά του παραθύρου υπάρχουν δυνατότητες
βοήθειας, αλλαγής των βάσεων που διερευνούνται κλπ.
Μετά την είσοδο των όρων
αναζήτησης κάνετε κλικ στο search και περιμένετε τα αποτελέσματα της
αναζήτησης. Δίνονται ο αριθμός των επιτυχών αναζητήσεων και για κάθε αναζήτηση
η βάση στην οποία βρέθηκαν τα συγκεκριμένα στοιχεία. Κάθε αναφορά είναι
προσβάσιμη στην βάση με την λεπτομέρεια που αυτή προσφέρει (βιβλιογραφικά
στοιχεία, περίληψη κλπ.)
4. Οι τοπικές βάσεις δεδομένων
Α. AGRICOLA Database
Τι είναι η AGRICOLA Database: Ως μία από τις περισσότερο εμπεριστατωμένες πηγές
πληροφορίας των γεωπονικών και βιολογικών επιστημών των Η.Π.Α., η AGRICOLA (Agricultural Online Access) Database περιέχει βιβλιογραφικά
δεδομένα για κείμενα πού έχει προμηθευτεί η National Agricultural Library (NAL) του U.S. Department of Agriculture (USDA). Ολόκληρη η β.δ. από
το 1970 περιέχει 3,600,000 αναφορές σε άρθρα περιοδικών, μονογραφίες, θέσεις,
ευρεσιτεχνίες, λογισμικό, οπτικοακουστικά μέσα και τεχνικές αναφορές που
σχετίζονται με τις γεωπονικές επιστήμες. Η AGRICOLA χρησιμεύει για τον εντοπισμό άρθρων και βιβλιογραφικό έλεγχο της συλλογής
της NAL. Η β.δ. καλύπτει επισταμένα νέες
εκδόσεις παγκοσμίως σε γεωπονικά και σχετικά αντικείμενα. Η AGRICOLA ανανεώνεται μηνιαία.
Ποιός πρέπει να χρησιμοποιεί την AGRICOLA Database και γιατί:
Οποιοσδήποτε χρειάζεται πρόσβαση σε πληροφορίες σχετικές με την γεωπονία για
τον εντοπισμό βιβλιογραφίας από περιοδικά και μονογραφίες από τις Η.Π.Α. και
τον υπόλοιπο κόσμο.
Η θεματολογία της AGRICOLA περιέχει:
|
Agricultural
Products
|
Feed
Science
|
Natural
Resources
|
|
Animal
Science
|
Fertilizers
|
Pesticides
|
|
Aquaculture
|
Fibers
and Textiles
|
Physiology
|
|
Biotechnology
|
Food and
Nutrition
|
Plant and
Animal
|
|
Botany
|
Forestry
|
Plant
Sciences
|
|
Conservation
|
Horticulture
|
Pollution
|
|
Cytology
|
Human
Ecology
|
Public
Health
|
|
Agricultural
Economics
|
Human
Nutrition
|
Rural
Sociology
|
|
Agricultural
Education
|
Hydrology
|
Soil
Sciences
|
|
Energy
|
Hydroponics
|
Veterinary
Medicine
|
|
Engineering
|
Microbiology
|
Water
Quality
|
Β. AGRIS Database
Τι είναι η AGRIS Database: Αυτή η διεθνής βάση δεδομένων περιέχει περισσότερο από 1
εκατομμύριο αρχεία και είναι σχεδιασμένη για να βοηθά τον εντοπισμό πληροφορίας
που σχετίζεται με την παγκόσμια παραγωγή τροφής. Η AGRIS, που είναι το αντίστοιχο
της έντυπης έκδοσης AgrIndex, παράγεται από τον
Παγκόσμιο οργανισμό Τροφής και Γεωργίας ( Food and Agriculture Organization (FAO)) των Ηνωμένων Εθνών.
Ποιά είναι η πηγή της AGRIS Database: Πάνω από 100 εθνικά και
πολυεθνικά κέντρα παίρνουν μέρος στην συλλογή γεωπονικής βιβλιογραφίας από
περιοδικά, βιβλία, αναφορές, και πρακτικά συνεδρίων. Κάθε χώρα που συμμετέχει
στην AGRIS καταθέτει την πληροφορία που
δημοσιεύεται στην επικράτεια της. Όλη η πληροφορία προέρχεται από πηγές εκτός
Η.Π.Α. Ο FAO συντονίζει την ενέργεια με το
παγκόσμιο πληροφοριακό του σύστημα, διευκολύνοντας την ανταλλαγή γεωπονικής
πληροφορίας στις χώρες μέλη του. Η AGRIS είναι μία επεκτεταμμένη
παγκόσμια συλλογή γεωπονικής πληροφορίας, που αντικατοπτρίζει τα ερευνητικά
αποτελέσματα και την πληροφορία στην περιοχή της παραγωγής τροφής και γεωργικής
ανάπτυξης.
Η
θεματολογία της AGRIS περιέχει:
|
General
Agriculture
|
Forestry
|
|
Geography
and History
|
Animal
Production
|
|
Education
|
Aquatic
Science and Fisheries
|
|
Extension
and Advisory Work
|
Machinery
and Buildings
|
|
Administration
and Legislation
|
Natural
Resources
|
|
Ecomonics
|
Food
Science
|
|
Development
and Rural Sociology
|
Home
Economics
|
|
Plant
Production
|
Human
Nutrition
|
|
Protection
of Plants and Stored Products
|
Pollution
|
Γ.
Wilson Biological & Agricultural Index
Τα Wilson Βιολογικά και Γεωπονικά Περιεχόμενα (Wilson Biological & Agricultural Index )
προσφέρουν βιβλιογραφικές πληροφορίες για περισσότερα από 250 βασικά παγκόσμια
περιοδικά που δημοσιεύονται στην αγγλική γλώσσα στο χώρο των βιολογικών και
γεωπονικών επιστημών. Η βάση περιέχει επίσης αναφορές σε ανασκοπήσεις βιβλίων,
κεφάλαια και ετήσιες αναφορές, συμπληρώματα περιοδικών καί άρθρα. Βιογραφικά
και γεωπονικά περιεχόμενα προσφέρουν ευρύτητα στην κάλυψη της θεματολογίας και
σε σχετικά θέματα συμπεριλαμβανομένων των χημικών ουσιών, οικονομικών,
μηχανολογίας, ζωοτεχνίας, βιοχημείας, βιολογίας, βιοτεχνολογίας, βοτανικής,
κυτταρολογίας, οικολογίας, εντομολογίας, υδροβιολογίας, ιχθυολογίας,
δασοπονίας, δενδροκομίας, μικροβιολογίας, διατροφής, φυσιολογίας, εδαφολογίας,
κτηνιατρικής, και ζωολογίας.
|
Πηγή:
|
H.W.
Wilson Company, The
|
|
Κάλυψη:
|
1983-Παρόν
|
|
Αντίστοιχο έντυπο:
|
Biological
& Agricultural Index
|
|
Τύπος δεδομένων:
|
Bιβλιογραφικό
|
|
Αριθμός αρχείων:
|
542,000+
|
|
Ετήσια συμπληρ.:
|
56,700+
|
|
Θεματολογία:
|
- Agriculture
- Animal Science
- Biochemistry
- Biology
- Botany
- Cytology
- Ecology
- Engineering
- Entomology
|
- Environment
- Food & Agriculture
- Forestry
- Genetics
- Health Sciences
- Horticulture
- Life Sciences
- Marine Sciences
- Microbiology
|
- Nutrition
- Physiology
- Plant Genetics
- Public Health
- Research & Development
- Soils
- Veterinary Science
- Zoology
|
Δ.
Derwent Biotechnology Abstracts
Τα Derwent Biotechnology Abstracts προσφέρουν βιβλιογραφική κάλυψη
στα πλέον τρέχοντα και πρωτοποριακά επιστημονικά και τεχνικά ερευνητικά θέματα
στον χώρο της βιοτεχνολογίας, με έμφαση τις εμπορικές και βιομηχανικές
εφαρμογές. Τα Derwent Biotechnology Abstracts πριέχουν περιλήψεις από πλέον των
1,200 παγκοσμίων επιστημονικών και τεχνολογικών περιοδικών,
ευρεσιτεχνίες από 40 κέντρα ευρεσιτεχνίας, και παγκόσμιαα συνέδρια και
συναντήσεις. Περισσότερα από το 30% των αρχείων στην β.δ. Derwent Biotechnology Abstracts περιέχει πληροφορίες παγκοσμίων ευρεσιτεχνιών .
Η Derwent Biotechnology Abstracts καλύπτει επίσης γενετική
μηχανική, βιοχημική τεχνολογία, ζυμώσεις, κυτταροκαλιέργεια, και βιομηχανική
διαχείρηση αποβλήτων. Κάθε ένα από τα 200,000+ αρχεία περιέχουν τίτλους των
άρθρων των περιοδικών, συμπεριλαμβανομένου και του τίτλου του περιοδικού, έναν δευτερεύοντα βελτιωμένο τίτλο, ειδικά
διαμορφωμένους τίτλους για να καλύπτουν τις προόδους και τα περιεχόμενα νέων
ευρεσιτεχνιών, πλήρη βιβλιογραφικά δεδομένα και συμπαγείς περιλήψεις που
περιγράφουν την ερευνητική μεθοδολογία, τα αποτελέσματα και τα συμπεράσματα.
Επίσης περιλαμβάνονται τύποι και σχέδια.
|
Πηγή:
|
Derwent
Information Ltd.
|
|
|
|
|
Κάλυψη:
|
1982-Παρόν
|
|
Αντίστοιχο Έντυπο:
|
Derwent
Biotechnology Abstracts Journal
|
|
|
|
|
Τύπος δεδομένων:
|
Βιβλιογραφικά με περιλήψεις
|
|
Αριθμός αρχείων:
|
200,000+
|
|
Ετήσια συμπλήρ.:
|
16,000+
|
|
Θεματολογία:
|
- Biology
- Biotechnology
- Chemicals
- Food & Agriculture
|
- Genetics
- Health Sciences
- Pharmaceutical
- Plant Genetics
|
- Science & Technology
- Waste
|
Ε. ASFA (Aquatic Sciences and Fisheries Abstracts )
Η ASFA είναι
η β.δ. για τις επιστήμες που σχετίζονται με το νερό προσφέροντας πληροφορίες
για παγκόσμιες εξελίξεις στην επιστήμη, τεχνολογία και διαχείριση θαλάσσιου και
μή υδατικού περιβάλλοντος και οργανισμών.Η
ASFA παράγεται σε συνεργασία με τέσσερεις υπηρεσίες των Ηνωμένων Εθνών και ένα
αυξανόμενο δίκτυο εθνικών ερευνητικών κέντρων παγκοσμίως. Οι συμμετέχοντες από τα Η.Ε. είναι οι Food and Agriculture Organization, Intergovernmental
Oceanographic Commission, U.N. Division for Ocean Affairs and the Law of the
Sea, και το U.N. Environment
Programme.
Η ASFA προσφέρει περισσότερες
από 550,000 αναφορές και περιλήψεις από την παγκόσμια έρευνα στις
υδροεπιστήμες, συμπεριλαμβανομένων περιοδικών, πρακτικών συνεδρίων και άλλη
σχετική βιβλιογραφία. Η θεματολογία περιλαμβάνει διαχείριση ιχθύων,
πληθυσμιακές έρευνες, οικολογία, βιολογία και γενετική των υδρόβιων σπονδυλωτών
και μή, θαλάσσια βιοτεχνολογία, προϊόντα και την χρήση τους, λιμνολογία, φυσική
και χημική ωκεανογραφία, θαλάσσια γεωλογία, παρακολούθηση της μόλυνσης,
πρόβλεψη, έλεγχος και αποτέλεσμα στο περιβάλλον, συντήρηση, ανανέωση και
δημόσια υγεία.
Η ASFA καλύπτει την παρακάτω θεματολογία:
Biological Sciences and Living Resources; Ocean Technology, Policy and
Non-Living Resources; Aquatic Pollution and Environmental Quality; Aquaculture;
and Marine Biotechnology.
|
Πηγή:
|
Cambridge
Scientific Abstracts
Intergovernmental Oceanographic Commission
U.N. Environment Programme
U.N. Food and Agriculture Organization (FAO)
U.N. Division for Ocean Affairs and the Law of the Sea
|
|
Κάλυψη:
|
1978-Παρόν
|
|
Αντίστοιχο Έντυπο:
|
ASFA-1;
ASFA-2; ASFA-3; ASFA Aquaculture; ASFA Marine Biotechnology
|
|
Τύπος δεδομένων:
|
Βιβλιογραφικά με περιλήψεις
|
|
Αριθμός αρχείων:
|
550,000+
|
|
Ετήσια συμπλήρ:
|
36,000+
|
|
Θεματολογία:
|
- Aquaculture
- Biology
- Ecology
|
- Environment
- Marine Sciences
- Oceanography
|
- Pollution
- Science & Technology
- Water
|
Στ.
FSTA (Food Science and Technology Abstracts)
Η FSTA είναι η διεθνώς ανεγνωρισμένη β.δ. για τις επιστήμες και τεχνολογίες τροφίμων (Food Science and Technology Abstracts database) . Είναι σχεδιασμένη για τις ανάγκες όσων αναζητούν
ένα γρήγορη και αποδοτική πρόσβαση στην
παγκόσμια βιβλιογραφία των επιστημών και τεχνολογιών τροφίμων και ποτών.
Καλύπτει την επιστήμη τροφίμων, βρώσιμα προϊόντα και συσκευασία τροφίμων. Η β.δ.
περιέχει 550,000 βιβλιογραφικά αρχεία
από το 1969 μέχρι σήμερα. Η FSTA περιέχει πληροφορίες από παγκόσμια
επιστημονικά περιοδικά σχετικά με την επιστήμη και τεχνολογία τροφίμων, όπως
και πληροφορίες από ευρεσιτεχνίες. Βιβλία, πρακτικά συνεδρίων, αναφορές,
φυλλάδια, νομολογία κλπ. Όλες οι περιλήψεις είναι στα αγγλικά από υλικό που
αρχικά εκδίδεται σε 40 διαφορετικές γλώσσες.
|
Πηγή:
|
International
Food Information Service (IFIS)
|
|
Κάλυψη:
|
1969-Σήμερα
|
|
Αντίστοιχο Έντυπο:
|
Food
Science and Technology Abstracts (FSTA)
|
|
Τύπος δεδομένων:
|
Βιβλιογραφικά με περιλήψεις
|
|
Αριθμός αρχείων:
|
550,000+
|
|
Ετήσια συμπλήρ.:
|
20,000+
|
|
Θεματολογία:
|
- Beverages
- Biochemistry
- Biotechnology
- Dairy Science
- Food & Agriculture
- Government Regulations
|
- Health & Safety
- Health Sciences
- Marketing
- Microbiology
- Nutrition
- Packaging
|
- Pesticides
- Public Health
- R & D
- Science & Technology
- Toxicology
|
5. Άσκηση
Σε αυτή την
άσκηση θέλουμε να αναζητήσουμε βιβλιογραφικές πληροφορίες που αφορούν άρθρα για
θέματα γεωπονίας.
Αναζήτηση πληροφοριών για την μωσαϊκή του καπνού ή την διοξίνη στα κοτόπουλα
- Αρχικά θα επιλέξουμε τις βάσεις που θα συμπεριλάβουμε στην
αναζήτηση.
Ανακτήστε άρθρα που αφορούν την μωσαϊκή του
καπνού ή που σχετίζονται με την διοξίνη στα κοτόπουλα.
. Συνδεθείτε με την ιστοσελίδα
του εξυπηρετητή β.δ. του ΓΠΑ (http://bibws4:8590/?). Αφού
δώσετε τον κωδικό εισόδου επιλέξτε μία από τις
διαθέσιμες βάσεις. Εισάγετε τον
όρο αναζήτησης (π.χ. mosaic ή
dioxin) και πατήστε Enter. Για να
κάνετε την επιλογή σας πιό εντοπισμένη
χρησιμοποιήστε το build search και εισάγετε τις λέξεις (tobacco ή chicken) και (mosaic ή dioxin)
και πατήστε Enter. Κάνετε κλικ στο "search" για να δείτε λεπτομέρειες των σχετικών άρθρων
απο την Β.Δ.
Η αναζήτηση: tobaco or
mosaic τι επιτυγχάνει;
Υπάρχει κάποιο άρθρο σχετικό με το πρόσφατο πρόβλημα των διοξινών στην ανθρώπινη τροφική αλυσίδα;
Υπάρχουν διοξίνες στα ψάρια;
Περιεχόμενα
1) Η φιλοσοφία της βάσης
2) Η πρόσβαση στην PEST CABWeb .
3) Άσκηση
- Η φιλοσοφία της
βάσης
CAB INTERNATIONAL
Σύντομη
περιγραφή
Η CAB INTERNATIONAL (CABI) είναι ένας παγκόσμιος διακυβερνητικός που ιδρύθηκε το οργανισμός 1928.
Ανήκει στα κράτη μέλη που σήμερα αριθμούν τους 40. Ο οργανισμός είναι
οικονομικά αυτοδύναμος από την πώληση πληροφοριακών προϊόντων και υπηρεσιών,
προγραμμάτων έρευνας, παροχής συμβουλευτικών υπηρεσιών, εκπαιδεύσεις κ.λ.π. Η CABI έχει σκοπό την βελτίωση της ανθρώπινης διαβίωσης
παγκόσμια διαμέσου της διάδοσης, εφαρμογής και παραγωγής επιστημονικής γνώσης
σε υποστήριξη της ανάπτυξης, με έμφαση στην γεωπονία, δασοπονία, ανθρώπινη
υγεία και διαχείρηση φυσικών πόρων, με ιδιαίτερη έμφαση τις ανάγκες των
αναπτυσσόμενων χωρών. Περισσότερες πληροφορίες μπορούν να αντληθούν για την διαχείρηση ζιζανίων και φορέων ασθενειών και τους ευρύτερους σκοπούς της CAB INTERNATIONAL, όπως και για τις δράσεις της CABI, μέρος τις οποίες είναι και η έκδοση βιβλίων, περιοδικών
και ηλεκτρονικών προϊόντων
CABI και διαχείρηση ζιζανίων
Biological Science and Services
CAB INTERNATIONAL Βιολογικές Επιστήμες
και Υπηρεσίες συγκεντρώνονται σε δύο κέντρα:
Οι υπηρεσίες
διαχείρισης παρασίτων που παρέχονται είναι:
- αναγνώριση εχθρών
καλλιεργειών, παθογόνων, νηματοδών και παρασιτικών helminths
- εντοπισμός και διάγνωση
προβλημάτων ασθενειών φυτών
- βιοέλεγχος ειδών
παρασίτων που εισβάλλουν σε διατηρητέες περιοχές.
- Υποστήριξη για
ολοκληρωμένη αντιμετώπιση παρασίτων και βιολογικό έλεγχο στην γεωργία και
δασοπονία.
- Βιοσυστηματική έρευνα
σε αρθρόποδα, μικροοργανισμούς, νηματώδη, και helminths,
συμπεριλαμβανομένων και παρασίτων και ωφέλιμων οργανισμών.
- Εκπαίδευση,
συμβουλευτικές υπηρεσίες και συμβουλές σε όλες τις παραπάνω περιοχές.
2. Η πρόσβαση στην PEST
CABWeb
Η πρόσβαση στα
ηλεκτρονικά Abstracts των περιοδικών της CAB που προμηθεύεται σε έντυπη μορφή η Βιβλιοθήκη του ΓΠΑ
γίνεται από τον εξυπηρετητή PEST CABWeb στο site της CABI με την χρήση του
Netscape ή Explorer στην διεύθυνση (http://pest.cabweb.org) από οποιοδήποτε PC στο ΓΠΑ χωρίς την χρήση
ειδικών κωδικών εισόδου.
|
 PEST CABWeb PEST CABWeb
Καλύπτει εντομολογία, νηματολογία, παρασιτολογία, βιολογικός έλεγχος,
φυτοπαθολογία, και άλλα θέματα σχετικά με την διαχείρηση ζιζανίων. Το PEST CABWeb προσφέρει
ένα μοναδικό συνδυασμό επιστημονικής εξειδίκευσης και πληροφόρησης.
© 1998 CABI Publishing
Wallingford, Oxon, OX10 8DE, UK
Tel: +44 (0)1491 832111
Fax: +44 (0)1491 833508
|
 Bulletin of Entomological Research Bulletin of Entomological Research
 Biocontrol News and Information Biocontrol News and Information
 Nematological Abstracts Nematological Abstracts
 Review of Agricultural Entomology Review of Agricultural Entomology
 Review of Medical and Veterinary Review of Medical and Veterinary
Entomology
 Review of Plant Pathology Review of Plant Pathology
 Weed Abstracts Weed Abstracts
|
|

10 East 40th Street,
Suite 3203
New York, NY 10016, USA
Tel: +1 212 481 7018
Toll-free: 800 528 4841
Fax: 212 686 7993
|
 Crop Protection
Compendium Crop Protection
Compendium
Information and
Support
 Descriptions of Fungi and Bacteria Descriptions of Fungi and Bacteria
 Pest Identification Pest Identification
 Pest Management Research and Pest Management Research and
Training
 Calendar of Crop Protection Events Calendar of Crop Protection Events
 Herbicide Names - Common and Herbicide Names - Common and
Chemical
|
3. Άσκηση
Αναζητήσατε απο το Bulletin of Entomological Research τα abstracts των άρθρων του τεύχους Ιούνιος 1999 και επιλέξτε
αυτά που αφορούν διαφοροποιήσεις εντόμων με μοριακή βιολογία. Πόσα άρθρα
επιλέξατε;
Η βάση επιστημονικών περιοδικών του οίκου SWETS
Περιεχόμενα
1)
Ηλεκτρονική διεύθυνση , κωδικοί
εισόδου.
2)
Η φιλοσοφία της βάσης και τα περιοδικά
του οίκου.
3)
Αναζήτηση υλικού από τα επιλεγμένα
περιοδικά
α. Αναζήτηση με λέξεις κλειδιά
β. Πίνακας περιεχομένων
4)
Άσκηση
1. Ηλεκτρονική διεύθυνση , κωδικοί εισόδου.
Κωδικοί
χρηστών:
ΙI. Για την
βάση περιοδικών του οίκου SWETS (http://www.swetsnet.nl)
n
Τμήμα Γεωπονικής Βιοτεχνολογίας 8180006.biotech
Κωδικοί
εισόδου:
ΙI. Για την
βάση περιοδικών του οίκου SWETS (http://www.swetsnet.nl)
n
Τμήμα Γεωπονικής Βιοτεχνολογίας biotech11
2. Η φιλοσοφία της βάσης και τα περιοδικά του οίκου.
- Αναζήτηση
σε επίπεδο τίτλου άρθρου σε 15.000 επιστημονικά περιοδικά.
- Πρόσβαση σε
επίπεδο πλήρους κειμένου στα άρθρα 18 επιστημονικών περιοδικών (βλ.
κατάλογο) από το 1998 και μετά στα οποία είναι συνδρομήτρια η
Βιβλιοθήκη.
3. Αναζήτηση υλικού από τα επιλεγμένα περιοδικά
α. Αναζήτηση με λέξεις κλειδιά
β. Πίνακας περιεχομένων
4. Ασκηση
Παράρτημα Α
Κωδικοί
χρηστών:
Ι. Για την INSIDE της Βρετανικής Βιβλιοθήκης (http://inside.bl.uk:443)
n
Τμήμα Γεωπονικής Βιοτεχνολογίας biotech
ΙI. Για την
βάση περιοδικών του οίκου SWETS (http://www.swetsnet.nl)
n
Τμήμα Γεωπονικής Βιοτεχνολογίας 8180006.biotech
Κωδικοί
εισόδου:
Ι. Για την INSIDE της Βρετανικής Βιβλιοθήκης (http://inside.bl.uk:443)
n
Τμήμα Γεωπονικής Βιοτεχνολογίας biotech11
ΙI. Για την
βάση περιοδικών του οίκου SWETS (http://www.swetsnet.nl)
n
Τμήμα Γεωπονικής Βιοτεχνολογίας biotech11
Παράρτημα Β
Μερικές
χρήσιμες διευθύνσεις δικτύου που σχετίζονται με την βιβλιογραφική αναζήτηση
Library Associated Internet Sites
Ελληνικά πανεπιστήμια
www.aua.gr Γεωπονικό
Πανεπιστήμιο Αθηνών
www.ntua.gr Εθνικό
Μετσόβειο Πολυτεχνείο
Ευρωπαϊκή Ένωση
www.cordis.lu Βάση
δεδομένων CORDIS Ευρωπαική Ενωση
www.europa.eu.int Ευρωπαική
Ενωση
www.wau.nl/natura Natura
Διεθνής αναζήτηση βιβλιοθηκών
http://www.ohiolink.edu/cgi-bin/libweb-search.pl
Βιβλιοθήκες
www.lcweb.loc.gov Library of Congress
www.uspto.gov US
Patent Office
www.nalusda.gov US
National Agr. Library
www.bib.wau.nl/agralin/waulib.html Univ. of Wagenigen.Library
www.leeds.ac.uk/library/ Univ. of Leeds Library
www.bl.uk British
Library
grc.ntis.gov US
Gov. Research Center
Εκδότες
www.lights.com/publisher/ Διεθνής
αναζήτηση Εκδοτών
www.elsevier.nl Elsevier
Publisher
www.livanis.gr Εκδόσεις
Λιβάνη
www.macmillan.com Macmillan Publisher
Διεθνή περιοδικά
www.sciencemag.org Science
www.nature.com Nature
www.nationalgeographic.com National Geographic
www.pest.cabweb.org Pest CAB
Κεφάλαιο 2ο
Αναζήτηση και αποκομιδή δεδομένων από
μοριακές βιολογικές βάσεις δεδομένων
Εισαγωγή
Σε αυτή την
άσκηση θέλουμε να αναζητήσουμε πολλών ειδών πληροφορίες για βιολογικά μόρια (DNA και πρωτείνες). Θα
απαντήσουμε σε ερωτήσεις όπως:
- Τι
επιστημονικές δημοσιεύσεις σχετίζονται με το βιομόριο;
- Πως
θα βρούμε την αμινοξική ακολουθία; Τι ακολουθίες DNA αποκωδικοποιούν την
πρωτείνη;
- Πως
οργανώνονται τα γονίδια όσον αφορά εξόνια/ιντρόνια;
- Τι
γονίδια είναι σε επαφή με το γονίδιο που ενδιαφερόμαστε;
- Είναι
η τρισδιάστατη δομή της πρωτείνης γνωστή; Πως την βρίσκουμε;
- Η
πρωτείνη περιγράφεται σε βιβλιοθήκες γονιδιώματος;
- Ποιά
είναι η θέση του γονιδίου στο γένωμα; Σε ποιο χρωμόσωμα βρίσκεται το
γονίδιο
- Υπάρχουν
διαγνωστικοί δείκτες για πολυμορφισμούς;
- Υπάρχουν
γενετικές ασθένειες που συνδέονται με την πρωτείνη. Τι κλινικά συμπτώματα
έχουν;
Για να απευθύνουμε αυτές
τις ερωτήσεις θα χρησιμοποιήσουμε τα παρακάτω διαδικτυακά κέντρα (WWW sites)
και βάσεις δεδομένων που αυτά υποστηρίζουν.
Οι
βάσεις αυτές είναι προσβάσιμες μέσα από το διαδίκτυο σε εξυπηρετητές του
εξωτερικού. Οι βάσεις μπορούν να διερευνηθούν με ειδικό λογισμικό που
προσφέρεται και αναλύεται παρακάτω ή με λογισμικό πρόσβασης στο διαδίκτυο
(Internet) όπως Netscape Communicator ή Microsoft Explorer.
:
Για
παραδείγματα αναζήτησης θα εξετάσουμε δύο πρωτείνες. Το σωματίδιο αναγνώρισης
μηνύματος ( signal recognition particle (SRP)) και το ένζυμο μεταφεράση της φωσφοριβοσυλιομένης γουανίνης (hypoxanthine (guanine) phosphoribosyl transferase) που συχνά αναφέρεται ως
'hprt' ή 'hgprt'.
Α. Αναζήτηση πληροφοριών για την πρωτείνη σωματίδιο
αναγνώρισης μηνύματος (
signal recognition particle (SRP))
I. Με την
χρήση της Entrez
1.
Αρχικά θα χρησιμοποιήσουμε την βάση Entrez.
Η SRP έχει έξη
διαφορετικές πρωτεϊνικές υπομονάδες ή αλυσίδες. Ανακτήστε την ακολουθία της
ανθρώπινης SRP54 υπομονάδας. Συνδεθείτε με την ιστοσελίδα της Entrez (www.ncbi.nlm.nih.gov/Entrez). Επιλέξτε Proteins ώς την βάση πληροφοριών. Βεβαιωθείτε ότι η επιλογή "All Fields" και
Mode = "Automatic" είναι
επιλεγμένες. Εισάγετε τον όρο
αναζήτησης srp54 στην πρωτεϊνική Β.Δ. και πατήστε Enter. Για να κάνετε την επιλογή σας πιό
εντοπισμένη εισάγετε την λέξη human και πατήστε Enter. Κάνετε κλικ στο "Retrieve ... documents".
Μπορείτε να βρείτε την ανθρώπινη πρωτείνη στον κατάλογο (Η σωστή είναι P13624) ; Κάνετε κλικ στην "GenPept report" σύνδεση για αυτή την πρωτείνη για να δείτε λεπτομέρειες των σχετικών στοιχείων απο την Β.Δ. Πρώτα
θα δείτε γενικές πληροφορίες καιο στο τέλος την αμινοξική ακολουθία της
πρωτείνης.
Η Entrez μπορεί να σας οδηγήσει σε άλλα συναφή στοιχεία από
άλλες βάσεις δεδομένων. Κάνετε κλίκ στις συνδέσεις υπό τον τίτλο DBSOURCE (gi:...) για να δείτε αναφορές στην πυρηνική Β.Δ. Ένας άλλος τρόπος για να δείτε συνδέσεις σε άλλες β.δ.
είναι στην προηγούμενη σελίδα με τα ανακτώμενα κείμενα κάνετε κλίκ στο "Medline
link".
Στην αναζήτηση που κάνατε εισάγατε μία λέξη αναζήτησης
("srp54") μαι μετά επικεντρώσατε την αναζήτηση σας με μία άλλη λέξη ("human"). Είναι
δυνατόν να εισάγετε δύο λέξεις μαζί με τους παρακάτω τρεις τρόπους:
human srp54
human AND srp54
srp54 human
και να επιτύχετε το ίδιο
αποτέλεσμα. Δοκιμάστε έναν από αυτούς και συγκρίνατε τον με τα προηγούμενα αποτελέσματα σας.
Η αναζήτηση: srp54
OR human τι επιτυγχάνει?
Μπορείτε να κάνετε μία πιό εξειδικευμένη αναζήτηση με την επιλογή πεδίων. Πηγαίνετε
πίσω στην αρχική σελίδα αναζήτησης (www.ncbi.nlm.nih.gov/Entrez/protein.html). Επιλέξτε "Title word" αντί του "All fields". Εισάγετε srp54 ως όρο
αναζήτησης. Επικεντρώστε
την αναζήτηση σας επιλέγοντας το πεδίο "Organism"
και με όρο αναζήτησης human. Πρέπει να καταλήξετε με ένα μόνο στόχο την ανθρώπινη SRP54 πρωτείνη.
3.
Θέλουμε να βρούμε δημοσιεύσεις στην Medline που αναφέρονται σε πρωτείνες SR (που
εμπλέκονται στην ρύθμιση της επεξεργασίας του RNA ( regulation of RNA processing) και
έχουν δημοσιευθεί το 1997 ή 1998 στο
περιοδικό Nature, Cell ή Science. Για να το κάνετε αυτό επιλέξτε την
β.δ. PubMed. Εισάγετε "SR"
και "splicing" ώς
όρους αναζήτησης. Επικεντρώστε
την αναζήτηση σας με την επιλογή του πεδίου "Publication date" και εισάγετε
"1997
OR 1998" ως όρο αναζήτησης. Για περαιτέρω επικέντρωση επιλέξτε το πεδίο "Journal" και εισάγετε "Nature
OR Science OR Cell" ως όρο αναζήτησης.
Πόσα άρθρα βρήκατε;
Αυτή η αναζήτηση μπορεί να
γίνει κα με την έκφραση :
splicing [all
fields] AND sr [all fields] AND ( nature [JOUR] OR cell [JOUR] OR Science
[JOUR]) AND ( 1997 [publication date] OR 1998 [publication date])
3.
Ακολουθεί μία περισσότερο εξειδικευμένη αναζήτηση. Θέλουμε να βρούμε
πρωτεϊνικές ακολουθίες που έχουν μήκος ακολουθίας μεταξύ 10,000 και 20,000.
Πηγαίνετε στην πρωτεϊνική β.δ. , επιλέξτε το πεδίο "sequence length",
και εισάγετε "01000:02000"
(Τα αρχικά μηδενικά είναι
απαραίτητα γιατί οι όροι του μήκους της ακολουθίας είναι εξαψήφιοι ακέραιοι.
Εάν έχετε αμφιβολίες χρησιμοποιήστε "List terms" για να δείτε όλους τους όρους του καταλόγου. Ο
διαχειριστής περιοχής χρησιμοποιεί τους όρους με την σειρά και τον τύπο που
παρουσιάζονται στην αναζήτηση. )
Πόσες ακολουθίες βρήκατε;
Κάνετε κλίκ σε μία από αυτές για να δείτε το ακριβή αριθμό αμινοξέων.
Β. Αναζήτηση πληροφοριών για το ένζυμο μεταφεράση
της φωσφοριβοσυλιομένης γουανίνης (hypoxanthine (guanine) phosphoribosyl transferase ή hprt ή hgprt)
Ι. Ακολουθία DNA
1. Πηγαίνετε στο NCBI Entrez και
επιλέξτε "Search WWW Entrez at NCBI Nucleotides ". Δοκιμάστε να βρείτε το
γονίδιο που κωδικοποιεί την ανθρώπινη hprt, δηλαδή ολόκληρη την γενομική ακολουθία
συμπεριλαμβανομένων των ιντρονίων. (Χρησιμοποιείστε ώς όρους αναζήτησης: hprt, human,
και complete. Κάνετε
κλικ στο κουμπί "Retrieve documents" να δείτε τα αποτελέσματα της έρευνας. Για να
δείτε επιμέρους ακολουθίες κάνετε κλικ στην επιλογή "Genbank report".
Εξετάσετε την περιοχή κειμένου της γενομικής ακολουθίας hprt και απαντήστε στις παρακάτω ερωτήσεις:
·
Ποιός είναι ο εισαγωγικός αριθμός (accession number) της
ακολουθίας ? (Ο πρώτος αριθμός στην γραμμή "ACCESSION"
)
·
Πόσα εξόνια υπάρχουν στο γονίδιο;
·
Πόσες μεταλλάξεις περιγράφονται και με τι ασθένειες
συνδέονται; Κρατείστε μία σημείωση των μεταλλάξεων και των σχετικών ασθενειών.
Όταν έχετε αποκομίσει την
ακολουθία του γονιδίου, πηγαίνετε στην προηγούμενη ιστοσελίδα και επιλέξτε
"Graphical view".Κάνετε κλικ σε ένα από τα εξόνια και επικεντρωθείτε στην περιοχή. Κατόπιν
πηγαίνετε πάλι πίσω και επιλέξτε "Protein link"
αντί "Graphical view". Επιλέξτε
"GenPept report".
·
Τι μέγεθος έχει η ανθρώπινη hprt (πόσα αμινοξέα);
ΙΙ. Πρωτεϊνική
ακολουθία
Πηγαίνετε πίσω στο NCBI-Entrez (www3.ncbi.nlm.nih.gov/Entrez/). Επιλέξτε "Proteins" ως β.δ. Προσπαθήστε να
βρείτε την hprt από Escherichia coli.
·
Πόσα αμινοξέα έχει η πρωτείνη;
·
Η πρωτείνη από E. coliέχει την ίδια συγγένεια υποστρώματος (substrate specificity)
όπως και η ανθρώπινη; (Εξετάστε τα αποτελέσματα που αφορούν την E. coli HPRT και κυττάξτε την περιοχή κειμένου στα σχόλια "Comment ...")
·
Σε ποιές επιστημονικές δημοσιεύσεις αναφέρεται η
ακολουθία; (Εξετάστε συνδέσεις στην MEDLINE)
.
Πηγαίνετε πίσω στο NCBI-Entrez (www3.ncbi.nlm.nih.gov/Entrez/). Επιλέξτε "Proteins" ως β.δ. Προσπαθήστε να
βρείτε την hprt από Bacillus subtilis.
ΙII. Γενομικά δεδομένα
1. Το ανθρώπινο γένωμα.
Για αυτές τις ασκήσεις θα
χρησιμοποιήσουμε την β.δ. GDB.
Αναζητήστε πληροφορίες για την HPRT. Από την
αρχική σελίδα της GDB επιλέξτε "All biological data" και "keyword"
και χρησιμοποιήστε τον όρο "hypoxanthine" για την αναζήτηση σας .
·
Ποιά γονίδια κωδικοποιούν την HPRT?
·
Ποιό από αυτά τα γονίδια έχει ένα συναφές φαινότυπο.
(συνδέεται με κάποια ασθένεια);
·
Σε ποιό χρωμόσωμα είναι αυτό το γονίδιο; Σε ποια
θέση; Προσπαθήστε να βρείτε ένα χάρτη
του χρωμοσώματος που να δείχνει την θέση της hprt.
·
Ποιά γονίδια βρίσκονται εκατέρωθεν αυτού της HPRT?
·
Υπάρχουν ζευγάρια εκκινητών για την ενίσχυση
ολόκληρης της περιοχής κωδικοποίησης της HPRT ;
·
Τι ασθένειες σχετίζονται με την HPRT ? Τι κλινικά συμπτώματα έχουν;
2.
Το γένωμα της E. coli .
Το πλήρες γένωμα της E. coli είναι διαθέσιμο από το 1997. Επιλέξτε στην NCBI Entrez "Search the NCBI genomes database" και πηγαίνετε στο γένωμα της E. coli . Προσπαθήστε να εντοπίσετε το γονίδιο της HPRT στην πυρηνική θέση ~142,000.
·
Τι γονίδια βρίσκονται κοντά στο γονίδιο της HPRT?
Κεφάλαιο 3ο
Ανάζήτηση
ακολουθιών πυρηνικών οξέων χρησιμοποιώντας το Internet
Εισαγωγή
Σε αυτή την
άσκηση θέλουμε να αναζητήσουμε πολλών ειδών πληροφορίες για βιολογικά μόρια (DNA και πρωτείνες). Θα
απαντήσουμε σε ερωτήσεις όπως:
- Πως
θα βρούμε την αμινοξική ακολουθία; Τι ακολουθίες DNA αποκωδικοποιούν την
πρωτείνη;
- Πως
οργανώνονται τα γονίδια όσον αφορά εξόνια/ιντρόνια;
- Τι
γονίδια είναι σε επαφή με το γονίδιο που ενδιαφερόμαστε;
- Ποιά
είναι η θέση του γονιδίου στο γένωμα; Σε ποιο χρωμόσωμα βρίσκεται το
γονίδιο
Για να απευθύνουμε αυτές
τις ερωτήσεις θα χρησιμοποιήσουμε τα παρακάτω διαδικτυακά κέντρα (WWW sites)
και βάσεις δεδομένων που αυτά υποστηρίζουν.
Οι βάσεις
αυτές είναι προσβάσιμες μέσα από το διαδίκτυο σε εξυπηρετητές του εξωτερικού.
Οι βάσεις μπορούν να διερευνηθούν με ειδικό λογισμικό που προσφέρεται και
αναλύεται παρακάτω ή με λογισμικό πρόσβασης στο διαδίκτυο (Internet) όπως
Netscape Communicator ή Microsoft Explorer.
Η βάση
πυρηνικών ακολουθιών του EMBL (Ευρωπαϊκού Εργαστηρίου Μοριακής Βιολογίας)
Η βάση πυρηνικών ακολουθιών του EMBL είναι μία
πλήρης βάση ακολουθιών DNA και RNA που έχουν
συλλεχθεί από την επιστημονική
βιβλιογραφία, αιτήσεις για ευρεσιτεχνίες ή έχουν απευθείας σταλεί από ερευνητές
και ομάδες εντοπισμού ακολουθιών. Η συλλογή των δεδομένων γίνεται σε συνεργασία
με την GenBank (ΗΠΑ) και την DNA Database of Japan (DDBJ). Η βάση διπλασιάζεται σε μέγεθος κάθε 12 μήνες και τον Ιούνιο του
1997 αριθμούσε 931 εκατομμύρια βάσεις από 1432941 ακολουθίες.
Η βάση
πυρηνικών ακολουθιών GenBank
Η GenBank είναι η βάση γενετικών
ακολουθιών του NIH (Εθνικός Οργανισμός Υγείας των ΗΠΑ), μία συλλογή όλων
των δημοσιευμένων ακολουθιών DNA . Υπάρχουν περίπου
967,000,000 βάσεις σε 1,491,000 ακολουθίες τον Ιούνιο του 1997. Για παράδειγμα
μπορείτε να δείτε πληροφορίες για το γονίδιο της νευροινιδομάτώσης .Περισσότερες πληροφορίες για την τρέχουσα έκδοση
της GenBank είναι διαθέσιμες στο διαδίκτυο.
Μία καινούργια έκδοση είναι διαθέσιμη κάθε δύο μήνες.
Η GenBank είναι διαθέσιμη για
αναζήτηση μέσω της Entrez ή της SRS.
Ασκήσεις.
Α. Αναζήτηση πληροφοριών για την πρωτείνη σωματίδιο
αναγνώρισης μηνύματος (
signal recognition particle (SRP))
Αναζήτηση με την χρήση της SRS (Sequence Retrieval System)
1. Συνδεθείτε
με την SRS ιστοσελίδα (srs.ebi.ac.uk:5000).
Κάνετε κλίκ στο "Start a new SRS session". Επιλέξτε "Swissprot" ως β.δ.
και κάνετε κλικ στο "Continue". Στην
επόμενη σελίδα εισάγετε τον όρο "srp54"
σε ένα από τα πεδία
(AllText) και "human" σε ένα από τα άλλα πεδία
αναζήτησης. Κάνετε
κλίκ στο "Do query". Θα αποκομίσετε μία λίστα από SRP54 ακολουθίες, συμπεριλαμβανομένων και πρωτεϊνών που δεν είναι SRP54 . Για να κάνετε μία περισσότερο ακριβή αναζήτηση πηγαίνετε πίσω στην σελίδα αναζήτησης και επιλέξτε ως "Gene name" srp54. Σε
ένα άλλο πλαίσιο επιλέξτε ως "Organism" homo sapiens (η
μόνο homo). Το αποτέλεσμα είναι η ανθρώπινη SRP54 ακολουθία. Κάνετε κλικ στην επιλογή . Μπορείτε να βρείτε συνδέσεις στις πυρηνικές β.δ. ;
Ένας άλλος τρόπος για να
δείτε συνδέσεις με άλλες βάσεις είναι : Πατήστε το κουμπί "Link"
στο επάνω μέρος της σελίδας. Επιλέξτε "PROSITE" ως
βάση σύνδεσης (τσεκάροντας το κουτί στα αριστερά της λέξης "PROSITE" . Πατήστε "Continue".
Κάνετε κλικ στα αποτελέσματα από την PROSITE . Μια γραμμή της
PROSITE γράφει:
P-[LIVM]-x-[FYL]-[LIVMAT]-[GS]-x-[GS]-[EQ]-x(4)-[LIVMF].
Αυτή είναι η τυπική ακολουθία για SRP54 πρωτείνες.
2. Θέλουμε να βρούμε
πληροφορίες για πρωτείνες από E. coli που έχουν πληροφορίες για σύνδεση
με DNA στην περιοχή του κειμένου . Επιλέξτε Swissprot ως β.δ. Επιλέξτε στο πεδίο "FtKey" τον όρο dna_bind (προσέξτε τον χαρακτήρα underscore ! ) και εισάγετε στο πεδίο "Organism"
*coli. (Η έκφραση *coli σημαίνει κάθε λέξη του
τελειώνει σε 'coli'.)
3. Θέλουμε να βρούμε πρωτείνες της Escerichia coli που έχουν (πιθανά) διαμεμβρανικά τμήματα. Επιλέξτε Swissprot ως βάση
αναζήτησης. Επιλέξτε "FtKey" = "transmem" και "Organism" = "*coli".
4. Οπως στην προηγούμενη άσκηση με την β.δ. Entrez θέλουμε να βρούμε πρωτεϊνικές ακολουθίες με μήκος ακολουθίας μεταξύ 10,000 and 20,000. Στην SRS επιλέγετε Swissprot ως βάση, επιλέγετε το πεδίο "SeqLength" aκαι εισάγετε "10000:20000". Χρησιμοποιήστε άλλα νούμερα εάν δεν βρείτε κάτι. Βρίσκετε τα ίδια
αποτελέσματα με την Entrez?
Β. Αναζήτηση πληροφοριών για το ένζυμο μεταφεράση
της φωσφοριβοσυλιομένης γουανίνης (hypoxanthine (guanine)
phosphoribosyl transferase ή hprt ή hgprt)
Ι. Ακολουθία DNA
1. Συνδεθείτε
με την SRS ιστοσελίδα (srs.ebi.ac.uk:5000).
Κάνετε κλίκ στο "Start a new SRS session". Επιλέξτε "Swissprot" ως β.δ.
και κάνετε κλικ στο "Continue". Δοκιμάστε
να βρείτε το γονίδιο που κωδικοποιεί την ανθρώπινη hprt, δηλαδή
ολόκληρη την γενομική ακολουθία συμπεριλαμβανομένων των ιντρονίων.
(Χρησιμοποιείστε ώς όρους αναζήτησης: hprt, human, και complete. Εξετάσετε την περιοχή κειμένου της γενομικής ακολουθίας hprt και απαντήστε στις
παρακάτω ερωτήσεις:
·
Ποιός είναι ο εισαγωγικός αριθμός (accession number) της ακολουθίας ? (Ο
πρώτος αριθμός στην γραμμή " (AC)CESSION" )
·
Πόσα εξόνια υπάρχουν στο γονίδιο;
·
Τι μέγεθος έχει η ανθρώπινη hprt (πόσα αμινοξέα);
ΙΙ. Πρωτεϊνική
ακολουθία
1. Συνδεθείτε
με την SRS ιστοσελίδα (srs.ebi.ac.uk:5000).
Από την αρχική σελίδα της
SRS επιλέξτε
"Start a new SRS session".
Επιλέξτε Swissprot σαν β.δ.
και κάνετε κλικ στο "Continue".
Αναζητήστε την hprt πρωτείνη
από Bacillus subtilis. (Εισάγετε "Bacillus subtilis" στο πεδίο "Organism"
και "hprt" στο
πεδίο "All-text").
·
Κοιτάξετε το περιγραφικό τμήμα για την Bacillus hprt. Θα
βρείτε μία σύνδεση στην PROSITE, μία
πρωτεϊνική β.δ. που περιέχει πρωτεϊνικά μοτίφ. Ποιό είναι το χαρακτηριστικό
μοτίφ αυτής της ομάδος πρωτεϊνών; Πηγαίνετε πίσω στην SRS και επιλέξτε "Enzyme" ως β.δ. από αυτές των “Metabolic pathway” (αντί της Swissprot που επιλέξατε προηγουμένως). Αναζητήστε την "hypoxanthine".
·
Μπορείτε να βρείτε ένα άλλο ένζυμο που έχει την
υποξανθίνη (hypoxanthine) ως
υπόστρωμα.
Αναζητήστε την E. coli hprt
χρησιμοποιώντας την SRS και Swissprot ως β.δ.. (Organism:
Escherichia coli). Μπορείτε να αναζητήσετε ομόλογες πρωτεϊνες
χρησιμοποιώντας το πρόγραμμα αναζήτησης ακολουθιών BLAST .
Ζητείστε την παρουσίαση (View) σε μορφή FASTA και αντιγράψτε την ακολουθία με (cut) (κάνετε κλικ στο δεξί πλήκτρο του ποντικιού) και paste (κάνετε κλικ στο δεξί πλήκτρο του
ποντικιού) στο παράθυρο ακολουθίας του BLAST.
Ποια συναφή ένζυμα
στην E. coli hprt βρίσκετε
στο αποτέλεσμα της αναζήτησης από το ψάξιμο με το BLAST επιπλέον των ομολόγων της hprt από
άλλους οργανισμούς;
Γ.
Επιλογή ακολουθίας και μετάφραση.
I. Επιλογή ακολουθίας και μετάφραση.
Συνδεθείτε
με την SRS ιστοσελίδα (srs.ebi.ac.uk:5000).
Εντοπίστε την ακολουθία
ectrmd από την βάση EMBL
(File - Add Sequences from - Database). Εξετάστε τις λεπτομέρειες του αρχείου και βρείτε το ανοικτό πλαίσιο ανάγνωσης (ORF open reading frame) της
ριβοσωμικής πρωτείνης S16. Επιλέξτε
μόνον την ακολουθία του αποκωδικοποιείται η S16 (1771..2019) και κάνετε αποκοπή. Χρησιμοποιείστε το
πρόγραμμα μετάφρασης και κάνετε επικόλληση. Κατόπιν μεταφράστε την ακολουθία.
Χρησιμοποιώντας την β.δ. Swissprot αναζητήστε
την E. coli ribosomal protein S16 (rs16_ecoli). Συγκρίνατε
την με το αποτέλεσμα της μετάφρασης σας.
ΙΙ.
Μη κανονικός γενετικός κώδικας.
Επαναλάβατε
την μετάφραση της ακολουθίας X07479 από
την EMBL (a rat mitochondrial protein gene) με τον
κανονικό γενετικό κώδικα όπως επίσης και με τον μιτοχονδιακό κώδικα και
συγκρίνατε τα αποτελέσματα.
ΙΙΙ.
Σύγκριση πλαισίων ανάγνωσης ακολουθιών.
Το
τμήμα EST της EMBL περιέχει ακολουθίες με
περισσότερα λάθη από τα άλλα τμήματα της β.δ. Οι ερευνητές συχνά κάνουν έναν
έλεγχο στην βάση με την ακολουθία τους πρίν να την καταθέσουν. Και συχνά
αναφέρουν στα σχόλια την ομοιότητα με κάποια πρωτείνη της βάσης Swissprot.
Συνδεθείτε
με την SRS ιστοσελίδα (srs.ebi.ac.uk:5000).
Διερευνήστε το EST
τμήμα της EMBL (Seqrelated -> EMESTLIB )για εισαγωγές όπου ο ερευνητής αναφέρει ομοιότητα
με την cystic fibrosis protein.
Βρήτε
την ακολουθία που αναφέρεται και την αντίστοιχη ακολουθία από την Swissprot cftr_human.
Ευθυγραμίστε τις δύο ακολουθίες και
κάνετε την σύγκριση μεταξύ τους. Τι παρατηρείτε;
Κεφάλαιο 4ο
Ανάλυση
ακολουθιών πυρηνικών οξέων χρησιμοποιώντας το Internet
(Sequence analysis using Internet functions)
Η χρήση των γενετικών βάσεων δεδομένων για την δημιουργία χαρτών
περιοριστικών ενζύμων, μετάφραση και εύρεση ομολόγων ακολουθιών (με το BLAST).
Οι περισσότερες ασκήσεις που αναπτύσσονται παρακάτω χρησιμοποιούν την μέθοδο της αντιγραφής μιάς νουκλεοτιδικής ή πρωτεϊνικής ακολουθίας από παράθυρο (Netscape ή Explorer) σε παράθυρο. Γι’αυτό είναι χρήσιμο να χρησιμοποιούνται
δύο παράθυρα ταυτόχρονα με το ίδιο πρόγραμμα δικτύου (Netscape ή Explorer) και την μεταφορά της πληροφορίας με
«Αποκοπή-Επικόλληση» (Cut-Paste).
Διαδικτυακά κέντρα (Internet Sites) που θα χρησιμοποιηθούν:
Α. Δημιουργία χαρτών περιοριστικών
ενζύμων(Restriction mapping)
Συνδεθείτε με το NCBI Entrez στη διεύθυνση www.ncbi.nlm.nih.gov/Entrez . Ανακαλέστε
την νουκλεοτιδική ακολουθία με κωδικό αριθμό (accession code) X01405 στην
μορφή FASTA.
Αντιγράψτε την ακολουθία από την σελίδα του NCBI, και επικολήστε την στο παράθυρο ακολουθιών του Webcutter σε άλλο παράθυρο Netscape ή Explorer (www.medkem.gu.se/cutter). Κάνετε κλίκ στην επιλογή "Analyze
sequence" για
να δείτε τις θέσεις των περιοριστικών ενζύμων.
Τώρα θέλετε να εντοπίσετε
που “κόβει” το περιοριστικό ένζυμο BamHI.
Πηγαίνετε πίσω στην αρχική σελίδα του Webcutter με την επιλογή back του Netscape ή Explorer
and ακυρώστε
την επιλογή "Map of restriction sites". Επιλέξτε "Only the
following enzymes" και κάνετε κλικ στην επιλογή BamHI. Κάνετε κλίκ στο
"Analyze
sequence" για
να πάρετε τις θέσεις περιορισμού του BamHI .
Πόσες φορές και που κόβει το BamHI
την ακολουθία X01405;
Πηγαίνετε πίσω στην αρχική
σελίδα του Webcutter με την επιλογή back του Netscape ή
Explorer and επιλέξτε την επιλογή "All enzymes
in the database". Για να δείτε όλα τα
ένζυμα που κόβουν ακριβώς μία φορά μόνο την ακολουθία επιλέξτε "Enzymes cutting once". Κάνετε κλίκ στο "Analyze sequence" για να πάρετε τις
θέσεις περιορισμού. Ποιά ένζυμα κόβουν μία μόνο φορά την ακολουθία X01405 και σε ποιές θέσεις;
Β. Μετάφραση νουκλεοτιδικών
ακολουθιών σε αμινοξικές
Συνδεθείτε με το
NCBI Entrez στη διεύθυνση www.ncbi.nlm.nih.gov/Entrez
. Ανακαλέστε την νουκλεοτιδική
ακολουθία με κωδικό αριθμό (accession code) X01405 στην
μορφή FASTA. Για την μετάφραση της ακολουθίας θα
χρησιμοποιηθεί το πρόγραμμα στην
διεύθυνση www.medkem.gu.se/edu/translat.html .
Αντιγράψτε την ακολουθία X01405 όπως στην άσκηση με τα περιοριστικά ένζυμα στο αντίστοιχο παράθυρο της σελίδας
www.medkem.gu.se/edu/translat.html. Κάνετε κλίκ στην επιλογή "Translate"
για να δείτε τα τρία αναγνωστικά πλάισια. Προσπαθήστε να αναγνωρίσετε το
αναγνωστικό πλαίσιο (open reading
frame) που αντιστοιχεί στην ακολουθία p53. Μπορείτε να βοηθηθείτε από την περιγραφή της σελίδας NCBI για την Χ01405. Όταν έχετε αναγνωρίσει το σωστό αναγνωστικό πλαίσιο (1 από τα 3 δυνατά) κάνετε την μετάφραση πάλι αλλά επιλέξτε το σωστό αναγνωστικό πλαίσιο (με την επιλογή "Translate entire sequence and select reading
frame ...") ώστε να πάρετε την γραμμική αμινοξική ακολουθία που αντιστοιχεί στην p53 στο επάνω μέρος της σελίδας. Ποιό
είναι το σωστό αναγνωστικό πλαίσιο;
Γ. Εντοπισμός αμινοξικής
ακολουθίας από ηλεκτροφόρηση νουκλεοτιδικής .
|

|
1.
Έχετε καταφέρει να κλωνοποιήσετε ένα γονίδιο μιάς
άγνωστης πρωτείνης. Έχετε χρησιμοποιήσει την μέθοδο Sanger <Q>τερματισμού της αλυσίδας
με διδεοξυνουκλεοτίδια για τον εντοπισμό της ακολουθίας του DNA για έναν από τους
κλώνους σας. Ποιά είναι η ακολουθία που διαβάζετε από την ηλεκτροφόρηση;
2.
Έχοντας την ακολουθία DNA θέλουμε να εντοπίσουμε
ποιά πρωτείνη εκφράζει αυτό το γονίδιο. Υπάρχουν πιθανά δύο τρόποι
εντοπισμού:
Α. Εντοπίζοντας ομόλογες
ακολουθίες νουκλεϊνικών οξέων με την χρήση του BLAST και
Β. Μεταφράζοντας το τμήμα της
νουκλεοτιδικής ακολουθίας και εντοπίζοντας ομόλογες πρωτείνικές ακολουθίες με
την χρήση του BLAST .
Θα χρησιμοποιήσουμε την δεύτερη μέθοδο.
(Δοκιμάστε μόνοι σας την πρώτη). Για την μετάφραση της νουκλεοτιδικής ακολουθίας χρησιμοποιείστε τον μεταφραστή
στην σελίδα www.medkem.gu.se/edu/translat.html με την επιλογή "Translate" για να
δείτε τα τρία αναγνωστικά πλαίσια.
Μπορείτε να εντοπίσετε το σωστό αναγνωστικό πλαίσιο; (Βοηθεια: Δεν πρέπει να
περιέχει κωδικόνια τερματισμού ).
3.
Για να αναγνωρίσετε το σωστό αναγνωστικό πλαίσιο (open reading frame) που
αντιστοιχεί στην ακολουθία χρησιμοποιείστε το πρόγραμμα BLAST (όπως στην άσκηση 3) για
να εντοπίσετε ομόλογες ακολουθίες.
4.
Επαναλάβατε το 2Β και 3 για την συμπληρωματική
αλυσίδα του DNA. Ποιά
από τις έξη πιθανές πρωτεϊνικές ακολουθίες σας δίνει ομολογία με πραγματική
πρωτείνη. Ποιά είναι αυτή;
|
|
|
|
Μπορείτε να εντοπίσετε
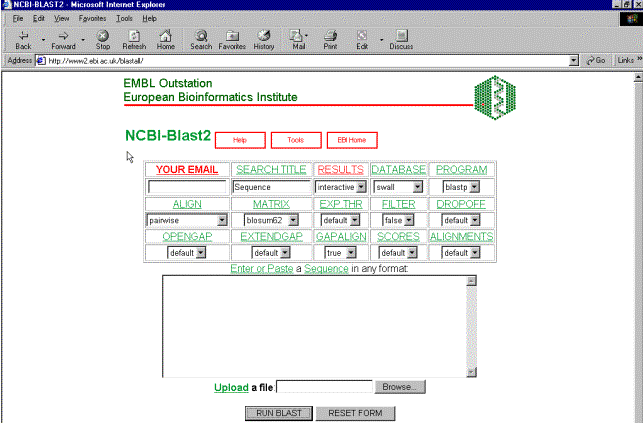
ομόλογες ακολουθίες χρησιμοποιώντας το πρόγραμμα αναζήτησης ακολουθιών BLAST (www2.ebi.ac.uk/blastall/). Eισάγετε
την ακολουθία σας στο παράθυρο (βλ. Σχήμα), επιλέγετε την DATABASE που θέλετε να ψάξετε και
κάνετε κλικ στο RUN
SEARCH. Μετά απο
λίγο θα δείτε τα αποτελέσματα της αναζήτησης. Για την περίπτωση πρωτεϊνικών
ακολουθιών επιλέξτε swissprot
για την DATABASE.

Κεφάλαιο 5
Ανάλυση
γενομικής πληροφορίας χρησιμοποιώντας το Internet
(Genomic information analysis using Internet functions)
Η χρήση των γενετικών βάσεων δεδομένων για την δημιουργία χαρτών
περιοριστικών ενζύμων, μετάφραση και εύρεση ομολόγων ακολουθιών (με το BLAST).
Οι περισσότερες ασκήσεις που αναπτύσσονται παρακάτω χρησιμοποιούν την μέθοδο της αντιγραφής μιάς νουκλεοτιδικής ή πρωτεϊνικής ακολουθίας από παράθυρο (Netscape ή Explorer) σε παράθυρο. Γι’αυτό είναι χρήσιμο να χρησιμοποιούνται
δύο παράθυρα ταυτόχρονα με το ίδιο πρόγραμμα δικτύου (Netscape ή Explorer) και την μεταφορά της πληροφορίας με
«Αποκοπή-Επικόλληση» (Cut-Paste).
Διαδικτυακά κέντρα (Internet Sites) που θα χρησιμοποιηθούν:
Δ. Ανάλυση της γενομικής πληροφορίας.
Οι παρακάτω ασκήσεις περιέχουν την ευθυγράμμιση 2-4 διαφορετικών ακολουθιών.
Χρήσιμες περιοχές του διαδικτύου για την ευθυγράμμιση 2 ακολουθιών είναι:
·
http://www.medkem.gu.se/ln/molbio/gene/msf.html (select global alignment!)
·
www2.ebi.ac.uk/clustalw
Για πολλαπλή ευθυγράμμιση:
·
http://www.medkem.gu.se/ln/molbio/gene/msf.html
1. Επαναλήψεις Alu .
Χρησιμοποιείστε την SRS για να ανακαλέσετε την νουκλεοτιδική ακολουθία Z82206, μία ακολουθία από το ανθρώπινο χρωμόσωμα 22 (Στην SRS επιλέξτε EMBL ως βάση δεδομένων και εισάγετε τον κωδικό στο πεδίο
"AccNumber" . Η περιοχή επεξηγήσεων της ακολουθίας μπορεί να χρησιμοποιηθεί για την αναγνώριση των επαναλήψεων Alu ("repeat region .. Alu.."). Μέρος του πίνακα στοιχείων της περιοχής επεξηγήσεων θα είναι όπως παρακάτω:
FT
repeat_region 1495..1797
FT /note="AluSp repeat: matches 1..303 of consensus"
FT
repeat_region 2171..2375
FT /note="AluJo repeat: matches 100..302 of
consensus"
FT /note="incomplete repeat"
FT
repeat_region 2443..2519
FT /note="MIR repeat: matches 191..260 of consensus"
FT
repeat_region 2664..2966
FT /note="AluSp repeat: matches 1..303 of
consensus"
FT
repeat_region 3146..4279
FT /note="TIGGER1 repeat: matches 2418..1298
of consensus"
FT
repeat_region 4273..5430
FT /note="TIGGER1 repeat: matches 1172..1 of
consensus"
FT
repeat_region 6761..7062
FT /note="AluSx repeat: matches 1..302 of
consensus"
FT
repeat_region 7771..8069
FT
/note="AluSc
repeat: matches 1..299 of consensus"
FT
repeat_region 8289..8692
FT /note="MLT1A1 repeat: matches 3..365 of
consen
Χρησιμοποιήστε πολλαπλή
ευθυγράμμιση (www.medkem.gu.se/edu/msf.html) για
να συγκρίνετε τέσσερεις διαφορετικές επαναλήψεις Alu. Επιλέξτε για παράδειγμα τις περιοχές 1495..1797, 2171..2375, 6761..7062 and 7771..8069. Είναι
ομόλογες;
2. Δομή εξονίων - ιντρονίων.
- Αναγνώριση γονιδίου με το BLAST. Χρησιμοποιείστε την ίδια ακολουθία με κωδικό Z82206 όπως και παραπάνω. Στην
περιοχή επεξήγησης υπάρχει πληροφορία για ένα τμήμα της το (<20814..21617). Χρησιμοποιείστε
το BLAST
(www.ncbi.nlm.nih.gov/cgi-bin/BLAST/nph-psi-blast?Jform=1) για να συγκρίνετε την ακολουθία αυτού του τμήματος με την βάση
δεδομένων. Επιλέξτε το πρόγραμμα blastn
και ως βάση την nr). Ποιά
είναι η πρωτείνη που κωδικοποιείται από το εξόνιο;
- Ευθυγράμμιση γενομικής
ακολουθίας με mRNA. Ανακαλέστε τις ακολουθίες DNA με κωδικούς
V00594 (Human mRNA για την
metallothionein) και J00271 (αντίστοιχη γενομική ακολουθία). Συγκρίνετε αυτές τις
ακολουθίες με ευθυγράμμιση ακολουθιών. Χρησιμοποιείστε το (www.medkem.gu.se/edu/msf.html) . Βασιστείτε στην ευθυγράμμιση για να πείτε πόσα εξόνια υπάρχουν στο γονίδιο. Συγκρίνετε τα
αποτελέσματα σας με την επεξήγηση που δίνεται στην περιγραφή του J00271.

