Άσκηση 4
Ανάλυση
ακολουθιών πυρηνικών οξέων χρησιμοποιώντας το Internet
(Sequence analysis using Internet functions)
Η χρήση των γενετικών βάσεων δεδομένων για την δημιουργία
χαρτών περιοριστικών ενζύμων, μετάφραση και εύρεση ομολόγων ακολουθιών (με το BLAST).
Οι περισσότερες ασκήσεις που αναπτύσσονται παρακάτω χρησιμοποιούν την μέθοδο της αντιγραφής μιάς νουκλεοτιδικής ή πρωτεϊνικής ακολουθίας από παράθυρο (Netscape ή Explorer) σε παράθυρο. Γι’αυτό είναι χρήσιμο να χρησιμοποιούνται
δύο παράθυρα ταυτόχρονα με το ίδιο πρόγραμμα δικτύου (Netscape ή Explorer) και την μεταφορά της πληροφορίας με
«Αποκοπή-Επικόλληση» (Cut-Paste).
Διαδικτυακά κέντρα (Internet Sites) που θα χρησιμοποιηθούν:
- NCBI Entrez
(http://www.ncbi.nlm.nih.gov/Entrez)
- SRS (Sequence retrieval system) ( http://srs.ebi.ac.uk )
- (http://www.firstmarket.com/cutter/cut2.html) Webcutter (για τον εντοπισμό
θέσεων περιοριστικών ενζύμων)
- Sequence translator at ebi (http://www2.ebi.ac.uk/translate
)
- BLAST για
αναζήτηση ακολουθιών (www2.ebi.ac..uk/blastall)
- CLUSTALW για ευθυγράμμιση ακολουθιών (www2.ebi.ac.uk/clustalw/)
- bio.lundberg.gu.se/edu/translat.html
για μετάφραση νουκλεοτιδικών σε αμινοξικές ακολουθίες
- bio.lundberg.gu.se/edu/msf1.html
για ευθυγράμμιση δύο ακολουθιών
- bio.lundberg.gu.se/edu/msf2.html
για πολλαπλή ευθυγράμιση ακολουθιών
Α. Δημιουργία χαρτών περιοριστικών
ενζύμων(Restriction mapping)
Συνδεθείτε με το NCBI Entrez στη διεύθυνση http://www.ncbi.nlm.nih.gov/Entrez
. Ανακαλέστε την νουκλεοτιδική
ακολουθία με κωδικό αριθμό (accession code) X01405 στην
μορφή FASTA.
Αντιγράψτε την ακολουθία από την σελίδα του NCBI, και επικολήστε την στο παράθυρο ακολουθιών του Webcutter σε άλλο παράθυρο Netscape ή Explorer (http://www.firstmarket.com/cutter/cut2.html) .
Κάνετε κλίκ στην επιλογή "Analyze sequence" για να δείτε τις
θέσεις των περιοριστικών ενζύμων.
Τώρα θέλετε να εντοπίσετε
που “κόβει” το περιοριστικό ένζυμο BamHI.
Πηγαίνετε πίσω στην αρχική σελίδα του Webcutter με την επιλογή back του Netscape ή Explorer
and ακυρώστε
την επιλογή "Map of restriction sites". Επιλέξτε "Only the
following enzymes" και κάνετε κλικ στην επιλογή BamHI. Κάνετε κλίκ στο
"Analyze
sequence" για
να πάρετε τις θέσεις περιορισμού του BamHI .
Πόσες φορές και που κόβει το BamHI
την ακολουθία X01405;
Πηγαίνετε πίσω στην αρχική
σελίδα του Webcutter με την επιλογή back του Netscape ή
Explorer and επιλέξτε την επιλογή "All enzymes
in the database". Για να δείτε όλα τα
ένζυμα που κόβουν ακριβώς μία φορά μόνο την ακολουθία επιλέξτε "Enzymes cutting once". Κάνετε κλίκ στο "Analyze sequence" για να πάρετε τις
θέσεις περιορισμού. Ποιά ένζυμα κόβουν μία μόνο φορά την ακολουθία X01405 και σε ποιές θέσεις;
Β. Μετάφραση νουκλεοτιδικών ακολουθιών σε
αμινοξικές
Συνδεθείτε με το
NCBI Entrez στη διεύθυνση www.ncbi.nlm.nih.gov/Entrez
. Ανακαλέστε την νουκλεοτιδική
ακολουθία με κωδικό αριθμό (accession code) X01405 στην
μορφή FASTA. Για την μετάφραση της ακολουθίας θα
χρησιμοποιηθεί το πρόγραμμα στην
διεύθυνση http://www2.ebi.ac.uk/translate.
Αντιγράψτε την ακολουθία X01405 όπως στην άσκηση με τα περιοριστικά ένζυμα στο αντίστοιχο παράθυρο της σελίδας http://www2.ebi.ac.uk/translate. Κάνετε κλίκ στην επιλογή "Translate"
για να δείτε τα τρία αναγνωστικά πλάισια. Προσπαθήστε να αναγνωρίσετε το
αναγνωστικό πλαίσιο (open reading
frame) που αντιστοιχεί στην ακολουθία p53. Μπορείτε να βοηθηθείτε από την περιγραφή της σελίδας NCBI για την Χ01405. Όταν έχετε αναγνωρίσει το σωστό αναγνωστικό πλαίσιο (1 από τα 3 δυνατά) κάνετε την μετάφραση πάλι αλλά επιλέξτε το σωστό αναγνωστικό πλαίσιο (με την επιλογή "Translate entire sequence and select reading
frame ...") ώστε να πάρετε την γραμμική αμινοξική ακολουθία που αντιστοιχεί στην p53 στο επάνω μέρος της σελίδας. Ποιό
είναι το σωστό αναγνωστικό πλαίσιο;
Γ. Εντοπισμός αμινοξικής
ακολουθίας από ηλεκτροφόρηση νουκλεοτιδικής .
|
|
1.
Έχετε καταφέρει να κλωνοποιήσετε ένα γονίδιο μιάς
άγνωστης πρωτείνης. Έχετε χρησιμοποιήσει την μέθοδο Sanger <Q>τερματισμού της αλυσίδας
με διδεοξυνουκλεοτίδια για τον εντοπισμό της ακολουθίας του DNA για έναν από τους
κλώνους σας. Ποιά είναι η ακολουθία που διαβάζετε από την ηλεκτροφόρηση; 2.
Έχοντας την ακολουθία DNA θέλουμε να εντοπίσουμε
ποιά πρωτείνη εκφράζει αυτό το γονίδιο. Υπάρχουν πιθανά δύο τρόποι
εντοπισμού: Α. Εντοπίζοντας ομόλογες ακολουθίες
νουκλεϊνικών οξέων με την χρήση του BLAST και Β. Μεταφράζοντας το τμήμα της
νουκλεοτιδικής ακολουθίας και εντοπίζοντας ομόλογες πρωτείνικές ακολουθίες με
την χρήση του BLAST . Θα χρησιμοποιήσουμε την δεύτερη μέθοδο.
(Δοκιμάστε μόνοι σας την πρώτη). Για την μετάφραση της νουκλεοτιδικής ακολουθίας χρησιμοποιείστε τον μεταφραστή
στην σελίδα http://www2.ebi.ac.uk/translate με την επιλογή "Translate" για να
δείτε τα τρία αναγνωστικά πλαίσια.
Μπορείτε να εντοπίσετε το σωστό αναγνωστικό πλαίσιο; (Βοηθεια: Δεν πρέπει να
περιέχει κωδικόνια τερματισμού ). 3. Για
να αναγνωρίσετε το σωστό αναγνωστικό πλαίσιο (open reading frame) που
αντιστοιχεί στην ακολουθία χρησιμοποιείστε το πρόγραμμα BLAST (όπως στην άσκηση 3) για
να εντοπίσετε ομόλογες ακολουθίες. 4. Επαναλάβατε
το 2Β και 3 για την συμπληρωματική αλυσίδα του DNA. Ποιά από τις έξη πιθανές
πρωτεϊνικές ακολουθίες σας δίνει ομολογία με πραγματική πρωτείνη. Ποιά είναι
αυτή; |
|
|
|
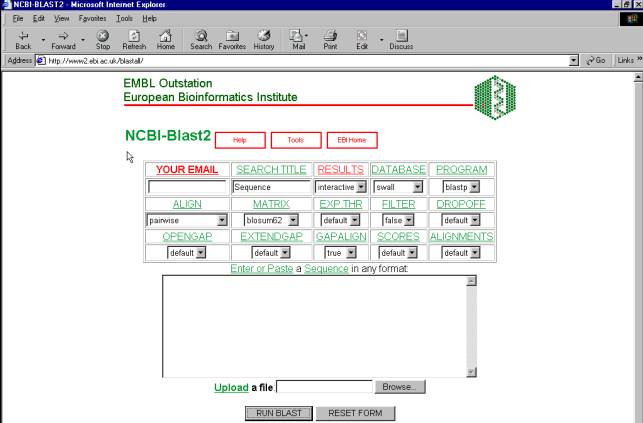
Μπορείτε να εντοπίσετε
ομόλογες ακολουθίες χρησιμοποιώντας το πρόγραμμα αναζήτησης ακολουθιών BLAST (www2.ebi.ac.uk/blastall/). Eισάγετε
την ακολουθία σας στο παράθυρο (βλ. Σχήμα), επιλέγετε την DATABASE που θέλετε να ψάξετε και
κάνετε κλικ στο RUN
SEARCH. Μετά απο
λίγο θα δείτε τα αποτελέσματα της αναζήτησης. Για την περίπτωση πρωτεϊνικών
ακολουθιών επιλέξτε swissprot
για την DATABASE.
![]()
ΠΑΡΑΡΤΗΜΑ
1
Α. Ο χάρτης περιοριστικών
ενζύμων της Χ01405
Enzyme Positions of Recognition
Sites Recognition Sequence
________
______________________________________ ___________
AatI 740 agg/cct
AccI 827 gt/mkac
AccIII 1762 t/ccgga
AflIII 408 a/crygt
AlwNI 423 cagnnn/ctg
AocI 369 cc/tna
.
.
Β. Μετάφραση
της Χ01405
Η γενομική
ακολουθία X01405
ID HSP53R standard; RNA; HUM; 2066 BP.
XX
AC X01405;XX
SV X01405.1XX
DT 07-NOV-1985 (Rel. 07, Created)DT 27-JAN-1995 (Rel. 42, Last updated, Version 8)XX
DE Human mRNA fragment for phosphoprotein p53XX
KW Alu repetitive sequence; antigen; phosphoprotein; tumor antigen.XX
OS Homo sapiens (human)OC Eukaryota; Metazoa; Chordata; Craniata; Vertebrata; Mammalia; Eutheria;OC Primates; Catarrhini; Hominidae; Homo.XX
RN [1]RP 1-2066RX MEDLINE; 85126934.
RA Matlashewski G., Lamb P., Pim D., Peacock J., Crawford L., Benchimol S.;RT "Isolation and characterization of a human p53 cDNA clone: expression ofRT the human p53 gene";RL EMBO J. 3:3257-3262(1984).XX
RN [2]RP 1-2066RA Crawford L.;RT ;RL Submitted (06-NOV-1985) to the EMBL/GenBank/DDBJ databases.XX
DR SWISS-PROT; P04637; P53_HUMAN.
DR TRANSFAC; T00671; T00671.
XX
CC Data kindly reviewed (06-NOV-1985)) by Crawford L.XX
FH Key Location/Qualifiers
FH
FT source 1..2066
FT /db_xref="taxon:9606"
FT /organism="Homo sapiens"FT CDS <1..883
FT /codon_start=2FT /db_xref="SWISS-PROT:P04637"
FT /product="p53"FT /protein_id="CAA25652.1"FT /translation="KTYQGSYGFRLGFLHSGTAKSVTCTYSPALNKMFCQLAKTCPVQL
FT WVDSTPPPGTRVRAMAIYKQSQHMTEVVRRCPHHERCSDSDGLAPPQHLIRVEGNLRVEFT YLDDRNTFRHSVVVPYEPPEVGSDCTTIHYNYMCNSSCMGGMNRRPILTIITLEDSSGNFT LLGRNSFEVRVCACPGRDRRTEEENLRKKGEPHHELPPGSTKRALPNNTSSSPQPKKKPFT LDGEYFTLQIRGRERFEMFRELNEALELKDAQAGKEPGGSRAHSSHLKSKKGQSTSRHKFT KLMFKTEGPDSD"FT old_sequence 1375..1379
FT /note="aacuu was au in [1]"FT /citation=[1]FT old_sequence 1392..1394
FT /note="ggu was gu in [1]"FT /citation=[1]FT misc_feature 1626..1936
FT /note="Alu-repeat sequence"FT misc_feature 2049..2054
FT /note="pot. polyA signal"FT polyA_site 2066
FT /note="polyA site"XX
SQ Sequence 2066 BP; 445 A; 568 C; 509 G; 544 T; 0 other; caaaacctac cagggcagct acggtttccg tctgggcttc ttgcattctg ggacagccaa 60 gtctgtgact tgcacgtact cccctgccct caacaagatg ttttgccaac tggccaagac 120 ctgccctgtg cagctgtggg ttgattccac acccccgccc ggcacccgcg tccgcgccat 180 ggccatctac aagcagtcac agcacatgac ggaggttgtg aggcgctgcc cccaccatga 240 gcgctgctca gatagcgatg gtctggcccc tcctcagcat cttatccgag tggaaggaaa 300 tttgcgtgtg gagtatttgg atgacagaaa cacttttcga catagtgtgg tggtgcccta 360 tgagccgcct gaggttggct ctgactgtac caccatccac tacaactaca tgtgtaacag 420 ttcctgcatg ggcggcatga accggaggcc catcctcacc atcatcacac tggaagactc 480 cagtggtaat ctactgggac ggaacagctt tgaggtgcgt gtttgtgcct gtcctgggag 540 agaccggcgc acagaggaag agaatctccg caagaaaggg gagcctcacc acgagctgcc 600 cccagggagc actaagcgag cactgcccaa caacaccagc tcctctcccc agccaaagaa 660 gaaaccactg gatggagaat atttcaccct tcagatccgt gggcgtgagc gcttcgagat 720 gttccgagag ctgaatgagg ccttggaact caaggatgcc caggctggga aggagccagg 780 ggggagcagg gctcactcca gccacctgaa gtccaaaaag ggtcagtcta cctcccgcca 840 taaaaaactc atgttcaaga cagaagggcc tgactcagac tgacattctc cacttcttgt 900 tccccactga cagcctccca cccccatctc tccctcccct gcgattttgg gttttgggtc 960 tttgaaccct tgcttgcaat aggtgtgcgt cagaagcacc caggacttcc atttgctttg 1020 tcccggggct ccactgaaca agttggcctg cactggtgtt ttgttgtggg gaggaggatg 1080 gggagtagga cataccagct tagattttaa ggtttttact gtgagggatg tttgggagat 1140 gtaagaaatg ttcttgcagt taagggttag tttacaatca gccacattct aggtaggggc 1200 ccacttcacc gtactaacca gggaagctgt ccctcactgt tgaattttct ctaacttcaa 1260 ggcccatatc tgtgaaatgc tggcatttgc acctacctca cagagtgcat tgtgagggtt 1320 aatgaaataa tgtacatctg gccttgaaac caccttttat tacatggggt ctagaacttg 1380 acccccttga gggtgcttgt tccctctccc tgttggtcgg tgggttggta gtttctacag 1440 ttgggcagct ggttaggtag agggagttgt caagtctctg ctggcccagc caaaccctgt 1500 ctgacaacct cttggtgaac cttagatcct aaaaggaaat gtcaccccat cccacaccct 1560 ggaggatttc atctcttgta tagatgatct ggatccacca agacttgttt tagctcaggg 1620 tccaatttct tttttctttt tttttttttt tttctttttc tttgagactg ggtctctttg 1680 ttgccccagg ctggagtgga gtggcgtgat ctggcttact gcagcctttg cctccccggc 1740 tcgagcagtc ctgcctcagc ctccggagta gctgggacca caggttcatg ccaccatggc 1800 cagccaactt ttgcatgttt tgtagagatg gggtctcaca gtgttgccca ggctggtctc 1860 aaactcctgg gctcaggcga tccacctgtc tcagcctccc agagtgctgg gattacaatt 1920 gtgagccacc acgtccagct ggaagggtca acatctttta cattctgcaa gcacatctgc 1980 attttcaccc cacccttccc ctcttctccc tttttatatc ccatttttat atcgatctct 2040 tattttacaa taaaactttg ctgcca 2066//
Η ακολουθία που θα δώση η σωστή μετάφραση θα είναι:
KTYQGSYGFRLGFLHSGTAKSVTCTYSPALNKMFCQLAKTCPVQLWVDST
50
PPPGTRVRAMAIYKQSQHMTEVVRRCPHHERCSDSDGLAPPQHLIRVEGN
100
LRVEYLDDRNTFRHSVVVPYEPPEVGSDCTTIHYNYMCNSSCMGGMNRRP
150
ILTIITLEDSSGNLLGRNSFEVRVCACPGRDRRTEEENLRKKGEPHHELP
200
PGSTKRALPNNTSSSPQPKKKPLDGEYFTLQIRGRERFEMFRELNEALEL
250
KDAQAGKEPGGSRAHSSHLKSKKGQSTSRHKKLMFKTEGPDSD
Γ. Εντοπισμός αμινοξικής
ακολουθίας από ηλεκτροφόρηση νουκλεοτιδικής .
Η ακολουθία που διαβάζεται από την ηλεκτροφόρηση είναι:
CTGCC CTGTG CAGCT GTGGG TTGAT TCCAC ACTC
Η μετάφραση της ακολουθίας DNA για την κανονική και την συμπληρωματική αλυσίδα δίνει τα παρακάτω πιθανά πρωτεϊνικά τμήματα:
Forward strand:
5' CUGCCCUGUGCAGCUGUGGGUUGAUUCCACACUC 3'
L P C A A V G * F H T
C P V Q L W V D S T L
A L C S C G L I P H
Reverse strand:
3' GACGGGACACGUCGACACCCAACUAAGGUGUGAG 5'
Q G T C S H T S E V S
A R H L Q P N I G C E
G Q A A T P Q N W V
